


ブタの形質とゲノム領域との相関を解析する際には、ゲノム全域にわたって高密度に配置された多型のあるDNAマーカーが必要です。また、形質を支配する遺伝子を探索する為に、他動物種との染色体との対応関係を考えるためには、遺伝子が高密度に染色体上に配置されていなければなりません。これまでにAGPでは、マイクロサテライトマーカーを中心とした多型マーカーとゲッチンゲンミニブタx梅山豚の実験家系を用いた連鎖地図の作製1や、大腸菌人工染色体(BAC)によるブタゲノムライブラリーを用いたマイクロサテライトマーカーの効率的な開発方法の確立(図1)2を行ってきました。
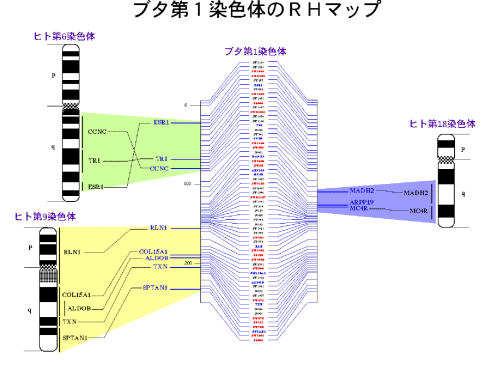
私たちは、放射線を照射したブタ大動脈内皮細胞と、チミジンキナーゼ欠損マウス細胞の融合細胞より作製した放射線雑種細胞(RH)パネルを用い、マイクロサテライトマーカーや発現遺伝子の断片配列の染色体上へのマッピングを行っています3,4。ブタ染色体上の遺伝子の位置がわかることにより、ヒト染色体の各部分との対応関係が明らかになり(図2)、形質の責任遺伝子の単離に役立つことが期待されます。これまでにRHパネルによって構築した染色体物理地図(RHマップ)は、私たちのデータベースを通じて検索することが可能です(図3)4。さらに、私たちはEST解析、完全長cDNA解読によって得られた配列情報から、発現遺伝子そのものの中5、あるいはイントロン配列の中から一塩基多型(SNP)を検出し(図4)、それらを用いてさらに信頼性の高い連鎖地図による遺伝子地図の作製を行うことを目指しています。
 |
 |
|
| 図1. BACクローンを用いたマイクロサテライトマーカーの開発法。詳細はAnim. Genet. 34:135-41, 2003を参照。 | 図2. ヒト-ブタ比較染色体地図。 | |
 |
 |
|
| 図3. RHマップの公開(SSRHデータベース)。 | 図4. 完全長cDNAの3'-UTR内でのSNP開発。 |
1Mikawa et al.: A linkage map of 243 DNA markers on swine resources familyconstructed
by intercross of Goettingen miniture pig and Meishan pig. Anim. Genet. 30:407-417, 1999.
2Fujishima-Kanaya et al.: Development of 50 gene-associated microsatellite
markers using BAC clones and the construction of a linkage map of swine
chromosome 4. Anim. Genet. 34:135-41, 2003.
3Hamasima et al.: Construction of a new porcine whole-genome framework map
using a radiation hybrid panel. Anim. Genet. 34:216-220, 2003.
4Mikawa et al.: Characterization of 298 ESTs from porcine back fat tissue
and their assignment to the radiation hybrid map. Mamm. Genome 15:315-22, 2004.
5Uenishi et al.: PEDE (Pig EST Data Explorer): construction of a database
for ESTs derived from porcine full-length cDNA libraries. Nucleic Acids Res. 32:D484-8, 2004.
![]()